SCPMA最新研究论文推荐!中国移动&玻色量子发布联合研究成果
玻色量子创始人&CEO文凯博士,上海交通大学特聘教授、药物化学与生物信息学中心主任张健为论文的通讯作者,这是玻色量子在“量子计算+生物制药”领域取得的首要研究突破,也建立了量子计算在生物制药领域实用化的重要里程碑。

分子对接是基于配体受体识别的锁钥模型提出的技术方法,通过计算配体受体之间的空间互补以及能量匹配来寻找其复合物模式,其在药物设计领域应用广泛,可分为刚性对接,半柔性对接以及柔性对接。传统的药物筛选是一个十分昂贵且需要消耗大量资源,一般需要花费数十亿美元,而成功率仅有10%。
近年来,随着功能强大的分子建模工具的发展以及蛋白质小分子复合物解析结构的数目的增长,基于结构的药物设计成为新药开发中广泛使用的路线之一。但传统模型的筛选方式耗时长,并且很难求得最优解,因此在先导化合物筛选中假阳性率较高,增加了药物研发成本。
以下为本篇论文的主要内容:
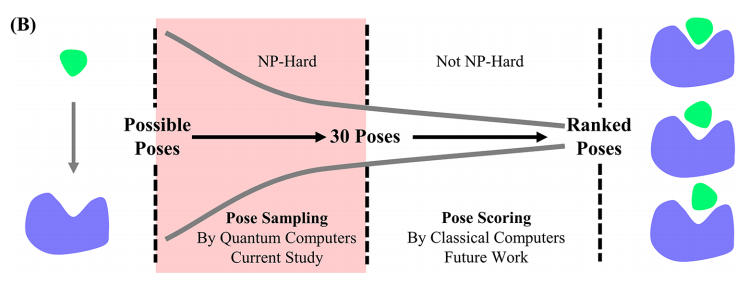
分子对接被广泛应用于虚拟筛选、先导化合物优化以及分子机制研究中,它通过空间采样来确定配体分子与受体蛋白间的结合姿态以及集合自由能(ΔGbind)。分子对接是药物发现的重要技术手段,分子对接过程的采样步骤是一个经典的NP-Hard 问题,其巨大的搜索空间和计算要求对于传统计算机充满了挑战。
在该研究中,联合研究团队提出了网格点匹配(Grid Point Matching,GPM)和原子特征匹配(Feature Atom Matching,FAM)算法模型,以便可以通过相干光量子计算机求解,从而将分子对接中的采样问题问题编码为QUBO(二次无约束二值优化)模型来加速采样过程。在CASF-2016数据集上测试显示GPM方法已经具备和经典的分子对接方法Glide SP相当的采样能力,在之前的研究中证实,相干光量子计算机的求解速度比传统计算机快1000倍,因此,该研究提出的算法模型可显著提升未来药物虚拟筛选效率和准确率。

图片2:(A)网格点匹配(GPM)和原子特征匹配(FAM)的工作流程。(B)该研究主要构建GPM和FAM方法加速分子对接中的采样过程。
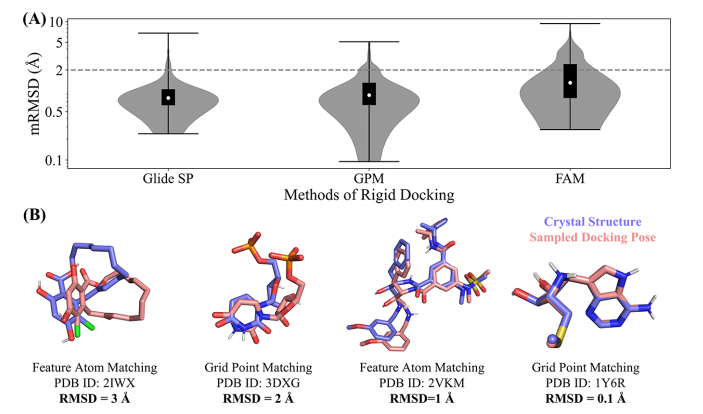
图3:FAM和GPM 模型表现和计算成本(A)三种采样方法Glide SP、GPM、FAM在测试集中的最小RMSD(minimum RMSD, mRMSD)分布。灰色虚线表示好的采样RMSD阈值。GPM代表“网格点匹配”,而FAM代表“原子特征匹配”。(B)FAM与GPM 方法采样得到的pose(红色表示) 与晶体结构中的pose (蓝色表示)比较。
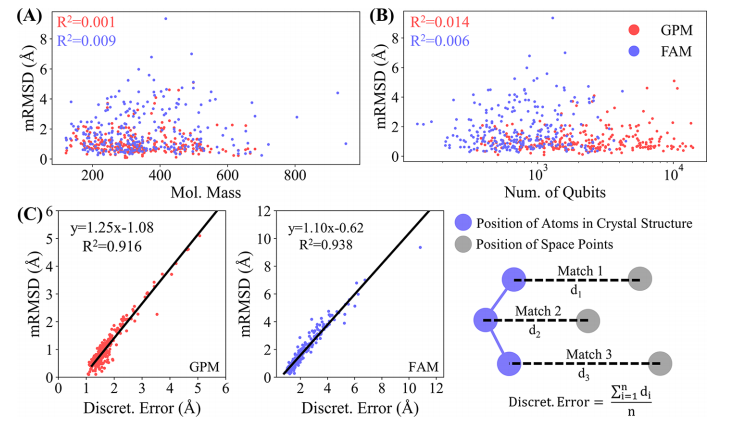
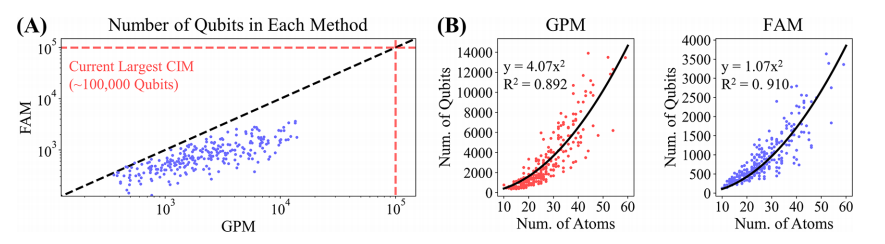
具体来讲,该研究提出的两个算法FAM和GPM 将分子对接过程的采样问题转化为配体原子和受体结合口袋的空间格点匹配问题,通过构建QUBO模型并在量子计算机上求得最优解,最后将求得的解转换为空间位置获得pose信息。通过在测试集上的大量验证表明,GPM 方法具有与经典采样方法Glide SP相当的采样能力。团队下一阶段将对FAM与GPM方法进行部署整合,补充打分步骤得到标准化的分子对接流程,并将其应用于类似ZINC的十亿级化合物数据库中执行虚拟筛选过程。
相干光量子计算机的技术特点使其可以构建全连接图,而其他量子计算机只能支持局部图的连接,这大大限制了其应用于实际场景过程。分子对接的取样过程是一个经典的NP-Hard问题,而近期研究表明相干光量子计算机对于求解该类问题具有巨大优势,该研究通过构建FAM和GPM方法并在相干光量子计算机上进行精确求解来解决分子对接中的采样问题。目前相干光量子计算机的最大求解规模远远超过了该算法模型在CASF数据集上应用所需的比特数,同时相干光量子计算机上的采样的运行时间仅以毫秒为单位,相较于经典计算机至少有3个数量级的提升,因此该研究提出的算法模型具有巨大的实际应用价值。
未来,玻色量子将携手更多合作伙伴持续进行“量子计算+生物制药”领域的深入研究技术突破和真机测试场景验证,共同研究量子计算在药物研发领域中的应用,涵盖虚拟筛选、蛋白质结构设计、分子动力学模拟等场景。



